install.packages("tidyverse")6 Programmer comme une pro
Une des premières choses à faire pour programmer comme une pro est de mettre son égo de côté. La grande majorité des programmeurs tapissent les murs de leurs cubicules d’aide-mémoires associés aux librairies de code qu’ils emploient. Je vous encourage donc fortement à imprimer les aide-mémoire associés aux libraries ggplot21 et dplyr2 et à les avoir à vos côtés au moment de vous lancer dans R.
6.1 La meta-librairie tidyverse
Jusqu’à maintenant, nous avons utilisé pour nos travaux une série de librairies différentes. Nous avons entre autres utilisé les librairies ggplot2, dplyr et tidyr. Si ces trois librairies travaillent si bien ensemble, c’est qu’elles font partie de la méta-librairie tidyverse. Cette dernière comprend une collection d’environ 25 librairies conçues par Hadley Wickham et ses collègues pour rendre le travail dans R plus facile, plus naturel.
Bien qu’elle soit une méta-librairie, tidyverse s’installe et s’active comme n’importe quelle librairie :
Cette opération pourrait prendre plusieurs minutes étant donné la quantité de librairies à installer.
N’utilisez la commande install.packages qu’une seule fois sur votre ordinateur. Lors des utilisations suivantes, vous n’avez qu’à l’activer avec la commande library.
library(tidyverse)── Attaching core tidyverse packages ──────────────────
✔ dplyr 1.1.4 ✔ readr 2.1.5
✔ forcats 1.0.0 ✔ stringr 1.5.1
✔ ggplot2 3.5.2 ✔ tibble 3.3.0
✔ lubridate 1.9.4 ✔ tidyr 1.3.1
✔ purrr 1.0.4
── Conflicts ───────────────── tidyverse_conflicts() ──
✖ dplyr::filter() masks stats::filter()
✖ dplyr::lag() masks stats::lag()
ℹ Use the conflicted package (<http://conflicted.r-lib.org/>) to force all conflicts to become errorsVous voyez dans ce message que l’activation de librairie tidyverse active automatiquement les librairies suivantes :
ggplot2dplyrtidyrtibblereadrforcatsstringrlubridate
Au terme de ce livre, nous aurons utilisé toutes ces librairies, à l’exception de stringr et de lubridate, qui servent respectivement à manipuler du texte et à manipuler des dates. Ces deux actions sont très communes, mais dépassent le cadre de ce livre.
Autrement dit, en activant la librairie tidyverse, vous avez automatiquement votre coffre à outils entier à portée de main.
Vous remarquerez peut-être aussi une deuxième section au message d’activation, soit celui avec liste des conflits. Un conflit existe lorsqu’une fonction dans une librairie porte le même nom qu’une fonction déjà présente dans votre environnement de travail. Et cela arrive fréquemment.
Dans le cas présent, on nous informe que la fonction filter de la librairie dplyr est venue cacher la fonction filter de la librairie stats. Le message nous informe ensuite que, si jamais nous voulons utiliser la fonction filter qui a été écrasée, on peut l’appeler en utilisant stats::filter. Autrement dit, le nom de la librairie, suivi de ::, suivi du nom de la fonction.
Cette façon de faire permet aussi d’utiliser une fonction dans une librairie sans avoir à activer toute la librairie au préalable.
Par exemple, pour enlever les lignes contenant des valeurs manquantes, il serait tout à fait légitime de faire :
tidyr::drop_na(tableau)plutôt que
library(tidyr)
drop_na(tableau)La métaphore des boîtes
Pour bien comprendre ces nuances, il faut vous imaginer les librairies de R comme des boîtes remplies d’outils.
La fonction install.packages est comme un camion d’Amazon qui vous livre la boîte d’outils. Il n’a besoin de livrer la boîte qu’une seule fois, ensuite, elle est chez vous pour toujours.
La fonction library prend le contenu d’une des boîtes et le vide sur votre établi. Tous les outils sont maintenant disponibles pour travailler.
Un conflit, c’est comme si en vidant le contenu d’une des boîtes, un des nouveaux outils se déposait par-dessus l’ancien. On peut encore accéder à l’ancien, mais si on ne fait rien de spécial, on attrape le nouveau qui est tombé par-dessus.
Enfin, la notation ::, permet de prendre un outil dans une des boîtes, de l’utiliser, et ensuite l’outil, attaché avec un élastique, retourne automatiquement dans sa boîte.
Les deux approches ont leurs avantages. Si vous avez besoin de beaucoup d’outils dans la boîte, ça peut être très pratique de vider toute la boîte (i.e. d’utiliser la fonction library). Mais si vous n’utilisez qu’un seul outil, une seule fois, peut-être que simplement utiliser l’outil et le retourner dans la boîte sera la meilleure chose à faire (i.e. utiliser :: sera la façon la plus efficace).
6.2 Labo : Densifier son code ggplot2
Une des choses à savoir à propos des programmeurs est qu’ils sont extrêmement paresseux. Eux vous diraient probablement efficaces, mais d’une façon ou d’une autre, ils détestent perdre leur temps à faire des choses répétitives et cherchent constamment des raccourcis pour accélérer leur travail.
En sachant quelques principes de R supplémentaires, le code R pour nos graphiques pourrait être grandement raccourci, et donc notre risque d’erreur grandement diminué.
La première chose à savoir est que les associations (mapping) n’ont pas besoin d’être répétés pour chaque couche graphique, pour autant qu’ils aient été spécifiés à l’appel original de la fonction ggplot. N’oubliez pas de commencer par charger la librairie palmerpenguins pour notre tableau de données :
library(palmerpenguins)
Attaching package: 'palmerpenguins'The following objects are masked from 'package:datasets':
penguins, penguins_rawEt ensuite, entrez le code suivant :
ggplot(data = penguins,
mapping = aes(x = body_mass_g, y = flipper_length_mm)) +
geom_point() +
geom_text(mapping = aes(label = species))Warning: Removed 2 rows containing missing values or values
outside the scale range (`geom_point()`).Warning: Removed 2 rows containing missing values or values
outside the scale range (`geom_text()`).
Premièrement, ce graphique est très laid! Ce n’est que pour donner un exemple…
Remarquez que le mapping de X et Y est maintenant dans la fonction ggplot plutôt que dans geom_point.
En travaillant ainsi, les variables body_mass_g et flipper_length_mm sont automatiquement associées aux coordonnées x et y pour toutes nos couches (geom_point et geom_text). Nous n’avons donc qu’à spécifier l’association supplémentaire dans notre couche de texte, où les étiquettes de nos données (label) proviendront de la variable species du tableau de données penguins.
Si jamais une propriété est mentionnée globalement et à l’intérieur d’une couche graphique, c’est l’association dans la couche graphique (dans le geom_) qui aura priorité pour cette couche en particulier.
L’autre notion importante à savoir pour densifier votre code encore plus est que dans R, le nom des arguments est optionnel, pour autant que l’on respecte l’ordre prescrit. Pour connaître cet ordre, il faut consulter l’aide de la fonction en question, p. ex.
?ggplotVous trouverez alors une ligne Usage qui devrait ressembler à ceci :
ggplot(data = NULL, mapping = aes(), ..., environment = parent.frame())
La section Usage de l’aide d’une fonction vous indique, entre autres, dans quel ordre R attend les arguments pour la fonction.
Si vous respectez cet ordre, vous n’avez pas besoin de nommer les arguments. Le code précédent pourrait donc être raccourci à ceci :
ggplot(penguins,aes(body_mass_g, flipper_length_mm)) +
geom_point() +
geom_text(aes(label = species))Warning: Removed 2 rows containing missing values or values
outside the scale range (`geom_point()`).Warning: Removed 2 rows containing missing values or values
outside the scale range (`geom_text()`).
Évidemment, personne ne vous force à utiliser cette façon de faire, mais elle est très commune et utilisée dans la plupart des exemples en ligne. Je l’utiliserais pour le reste des notes de cours.
6.3 Labo : Enchaîner les opérations de dplyr
Dans la préparation de vos données pour leur analyse, vous rencontrerez souvent (presque toujours en fait!) des situations où vous aurez plusieurs opérations à effectuer sur votre tableau de données avant qu’il ne soit prêt pour l’analyse.
On pourrait par exemple démarrer avec notre tableau de données sur les manchots, et vouloir obtenir au bout du compte un tableau contenant l’île et l’année de chacun des mâles Gentoo en ordre croissant de longueur d’aile.
Avec ce que l’on a vu jusqu’à présent, on aurait pu faire comme ceci :
a <- filter(penguins, sex == "male" & species == "Gentoo")
b <- arrange(a, flipper_length_mm)
select(b, island, year)# A tibble: 61 × 2
island year
<fct> <int>
1 Biscoe 2008
2 Biscoe 2009
3 Biscoe 2007
4 Biscoe 2009
5 Biscoe 2007
6 Biscoe 2007
7 Biscoe 2007
8 Biscoe 2007
9 Biscoe 2007
10 Biscoe 2008
# ℹ 51 more rowsVous constatez que pour y arriver, nous utilisons plusieurs objets intermédiaires (a et b) qui n’ont pas de fonction, autre que d’attendre l’opération suivante. L’utilisation de ces objets intermédiaires augmente aussi beaucoup le risque d’erreur.
Nous pourrions bêtement éliminer ces objets intermédiaires, comme ceci :
select(arrange(filter(penguins, sex == "male" & species == "Gentoo"), flipper_length_mm), island, year)Ce code accomplit la même tâche, mais vous serez sûrement d’accord, il est vraiment plus difficile à comprendre. Les problèmes sont nombreux. D’abord, le tableau de données duquel on démarre est caché quelque part au milieu du code. Ensuite, il faut lire du centre vers l’extérieur pour comprendre ce que le code fera, ce qui n’est pas très naturel.
Il existe dans R une solution à ce problème, que j’ai traduit comme l’opérateur d’enchaînement (|>, pipe operator). Le travail de l’opérateur d’enchaînement est de nous permettre d’écrire du code facile à lire pour nous, et lui s’occupe de le retraduire pour l’ordinateur.
Voyons comment nous aurions pu améliorer notre code à l’aide de cet opérateur, puis nous discuterons un peu de son fonctionnement.
penguins |>
filter(sex == "male" & species == "Gentoo") |>
arrange(flipper_length_mm) |>
select(island, year)Notre code est maintenant beaucoup plus facile à lire, puisque l’on peut maintenant lire normalement de haut en bas et de gauche à droite. Il décrit de façon beaucoup plus directe ce que l’on voulait faire : démarrer du tableau penguins, filtrer, trier et ensuite choisir des colonnes. Le nom du tableau de départ est toujours la première chose dans la chaîne, et ensuite les verbes décrivant nos opérations sont bien mis en évidence.
Si vous observez attentivement le bout de code précédent, vous verrez que pour chacun des verbes (fonctions), nous n’avons pas mentionné sur quel tableau de données travailler. C’est le travail de l’opérateur d’enchaînement : il prend ce qu’on lui fournit à sa gauche, et l’envoie comme premier argument de ce que l’on met à sa droite. On peut donc l’utiliser avec n’importe quelle fonction qui s’attend à recevoir un tableau de données comme premier argument, incluant ggplot :
penguins |>
filter(species == "Gentoo") |>
ggplot(aes(body_mass_g)) +
geom_histogram()`stat_bin()` using `bins = 30`. Pick better value with
`binwidth`.Warning: Removed 1 row containing non-finite outside the scale
range (`stat_bin()`).
Le code précédent nous fournit donc, de façon très compacte, un histogramme du poids des manchots Gentoo.
Notez la subtilité suivante : les opérations de dplyr s’enchaînent avec |> alors que les couches de ggplot2 s’ajoutent avec l’opérateur +. L’auteur de ces librairies s’excuse à plusieurs reprises dans son livre de cet imbroglio, mais la charge de travail pour établir la constance entre les deux librairies serait trop importante et briserait trop de code pré-existant.
La question qui apparaît dans plusieurs têtes à ce moment est souvent : wow, c’est fou, mais eh, est-ce que je pourrais faire toute mon analyse dans une seule chaîne? La réponse simple est : vous faites ce que vous voulez!
Mais, en général, on recommande de limiter la longueur d’une chaîne à 5-6 opérations qui ont un rapport entre elles. Si vous avez plus d’opérations que cela à faire, il peut être plus avantageux de séparer la chaîne en plusieurs morceaux et de conserver le résultat intermédiaire dans un objet. Particulièrement au moment du débogage, vous serez contents d’avoir travaillé de cette façon.
6.4 Labo : Grouper pour mieux résumer
Nous avons vu au Chapitre 4 qu’il existe une fonction summarize dans dplyr qui nous permet de résumer les données rapidement. Cette fonction est en fait beaucoup plus puissante qu’elle ne le paraissait si on lui combine un opérateur de regroupement. Rappelons-nous d’abord le fonctionnement de la fonction summarize (cette fois en utilisant aussi l’opérateur d’enchaînement) :
penguins |>
summarize(
poids_moyen = mean(body_mass_g, na.rm = TRUE),
ecart_type_poids = sd(body_mass_g, na.rm = TRUE)
)# A tibble: 1 × 2
poids_moyen ecart_type_poids
<dbl> <dbl>
1 4202. 802.Ce bout de code nous permet d’obtenir le poids moyen et l’écart-type du poids de manchots dans le tableau de données.
Si on ajoute l’opérateur de groupement, on pourrait obtenir ces chiffres par espèce :
penguins |>
group_by(species) |>
summarize(
poids_moyen = mean(body_mass_g, na.rm = TRUE),
ecart_type_poids = sd(body_mass_g, na.rm = TRUE)
)# A tibble: 3 × 3
species poids_moyen ecart_type_poids
<fct> <dbl> <dbl>
1 Adelie 3701. 459.
2 Chinstrap 3733. 384.
3 Gentoo 5076. 504.Ou même par espèce sur chacune des îles, etc.
penguins |>
group_by(species, island) |>
summarize(
poids_moyen = mean(body_mass_g, na.rm = TRUE),
ecart_type_poids = sd(body_mass_g, na.rm = TRUE)
)`summarise()` has grouped output by 'species'. You can
override using the `.groups` argument.# A tibble: 5 × 4
# Groups: species [3]
species island poids_moyen ecart_type_poids
<fct> <fct> <dbl> <dbl>
1 Adelie Biscoe 3710. 488.
2 Adelie Dream 3688. 455.
3 Adelie Torgersen 3706. 445.
4 Chinstrap Dream 3733. 384.
5 Gentoo Biscoe 5076. 504.Notez que l’objet retourné par summarize est lui aussi un tableau de données, qui peut être directement utilisé :
penguins |>
group_by(species, island) |>
summarize(
poids_moyen = mean(body_mass_g, na.rm = TRUE),
ecart_type_poids = sd(body_mass_g, na.rm = TRUE)
) |>
ggplot(aes(poids_moyen, ecart_type_poids)) +
geom_text(aes(label = species, color = island))`summarise()` has grouped output by 'species'. You can
override using the `.groups` argument.
C’est donc de cette façon que l’on pouvait obtenir les chiffres du tableau de contingence du Chapitre 3, avec le code suivant :
penguins |>
group_by(sex, species) |>
summarize(
n()
)`summarise()` has grouped output by 'sex'. You can
override using the `.groups` argument.# A tibble: 8 × 3
# Groups: sex [3]
sex species `n()`
<fct> <fct> <int>
1 female Adelie 73
2 female Chinstrap 34
3 female Gentoo 58
4 male Adelie 73
5 male Chinstrap 34
6 male Gentoo 61
7 <NA> Adelie 6
8 <NA> Gentoo 5Remarquez que pour ce cas particulier, il serait probablement plus efficace d’utiliser la fonction table de R de base :
table(penguins$sex, penguins$species)
Adelie Chinstrap Gentoo
female 73 34 58
male 73 34 616.5 Travailler avec des scripts
Vous avez probablement remarqué, particulièrement avec l’exemple où l’on a enchainé une chaîne de dplyr avec un graphique ggplot, qu’à mesure que notre code se complexifie, il devient de plus en plus ardu de construire nos commandes dans la petite ligne de la console de R. C’est pourquoi la majorité des gens qui travaillent avec R ne codent pas directement dans la console. Ils préparent plutôt des scripts (des séries de commandes) à l’aide d’un éditeur de code. Ils envoient ensuite leurs commandes déjà préparées à la console de R.
Dans RStudio, vous pouvez créer une nouvelle fenêtre de script à l’aide du menu File / New File / R Script. Votre environnement de RStudio se transformera alors comme ceci :
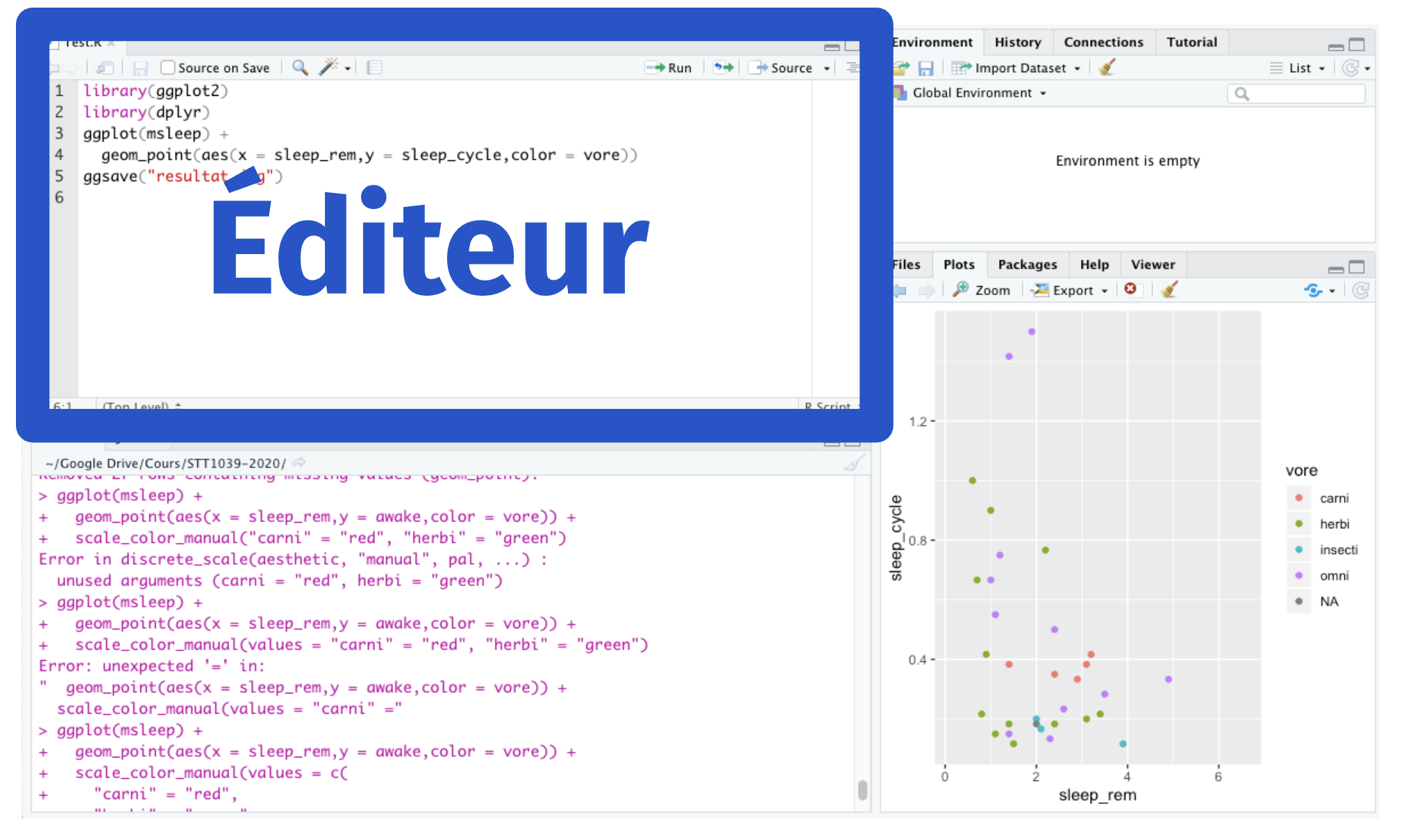
La partie de gauche sera séparée en deux, l’éditeur de code en haut, et la console de R en bas. Vous avez donc maintenant plein d’espace pour préparer vos lignes de code dans l’éditeur. Lorsqu’elles sont prêtes à envoyer à R, assurez que votre curseur est sur la bonne ligne et faites Ctrl+Enter (ou Cmd+Enter sur Mac) et RStudio enverra votre commande à la console. Outre le gain d’espace, l’avantage majeur à travailler de cette façon est que vous pouvez rassembler toutes les lignes de code associées à un projet, et sauvegarder ce script pour le réutiliser plus tard. Vous serez étonnés de voir le nombre de fois où un projet d’analyse s’étire à temps perdu sur plusieurs mois, voire même des années.
Une fois que vous travaillez de cette façon, il devient aussi intéressant d’insérer des lignes de commentaires dans votre code à l’aide du dièse ou hashtag (#). Toutes les lignes précédées d’un # seront considérées par R comme des commentaires et ne seront pas exécutées. Voici un petit exemple de script utilisant cette façon de faire :
# Chargement des librairies
library(tidyverse)
library(palmerpenguins)
# Création du premier graphique
ggplot(penguins) +
geom_point(aes(body_mass_g, flipper_length_mm))
# Sauvegarde du graphique
ggsave("resultat.jpg")Remarquez aussi que les lignes dans l’éditeur de code sont numérotées pour faciliter la discussion. Vous verrez peut-être parfois apparaître des avertissements (par exemple une bulle rouge avec un X) pour vous prévenir que cette ligne contient probablement des erreurs.
6.6 Le clavier est votre ami!
Une des choses que vous remarquerez si vous observez un programmeur travailler et qu’il touche rarement à la souris pendant qu’il code. La majorité du temps, ses doigts demeurent sur le clavier. Encore une fois, il s’agit d’une question d’efficacité. En connaissant quelques raccourcis clavier, votre travail gagnera en vitesse de façon spectaculaire.
Voici ceux que j’utilise le plus souvent :
| Raccourci | Windows | Mac |
|---|---|---|
Opérateur d’enchaînement (|>) |
Ctrl + Maj + M | Cmd + Maj + M |
| Opérateur d’assignation (<-) | Alt + - | Option + - |
| Lancer le code R du haut de la page jusqu’au curseur | Ctrl + Alt + B | Cmd + Option + B |
| Relancer une nouvelle fois le bloc de code que l’on vient d’exécuter | Ctrl + Maj + P | Alt + Cmd + P |
| Annuler la dernière action dans l’éditeur de code | Ctrl + Z | Cmd + Z |
Vous pouvez accéder à la liste complète des raccourcis de RStudio avec la commande Alt + Maj + K (Option + Maj + K sur Mac)
L’autre chose que je vous conseille fortement d’apprendre sur votre clavier est l’emplacement de certaines touches spéciales. Comme la plupart des langages de programmation, R utilise plusieurs caractères spéciaux pour définir certaines opérations. Ces caractères spéciaux sont rarement utilisés dans la vie de tous les jours et nous connaissons rarement leur emplacement sur nos claviers. Néanmoins, puisqu’ils reviennent fréquemment dans R, je vous conseille de les noter sur un post-it collé sur votre écran une fois que vous les aurez trouvés la première fois. Comme votre flow de pensée de programmation sera très fragile, particulièrement dans les débuts, il est préférable de ne pas perdre le fil chaque fois que vous aurez besoin d’écrire un caractère étrange.
Voici en rafale celles dont vous aurez besoin le plus souvent :
! # $ % ? & * ( ) [ ] ^ ~ < > | "
Notez que comme tous vos claviers sont différents, il m’est impossible ici de vous fournir la bonne façon pour vous d’écrire ces symboles sur votre ordinateur. C’est à vous de les trouver!